简介
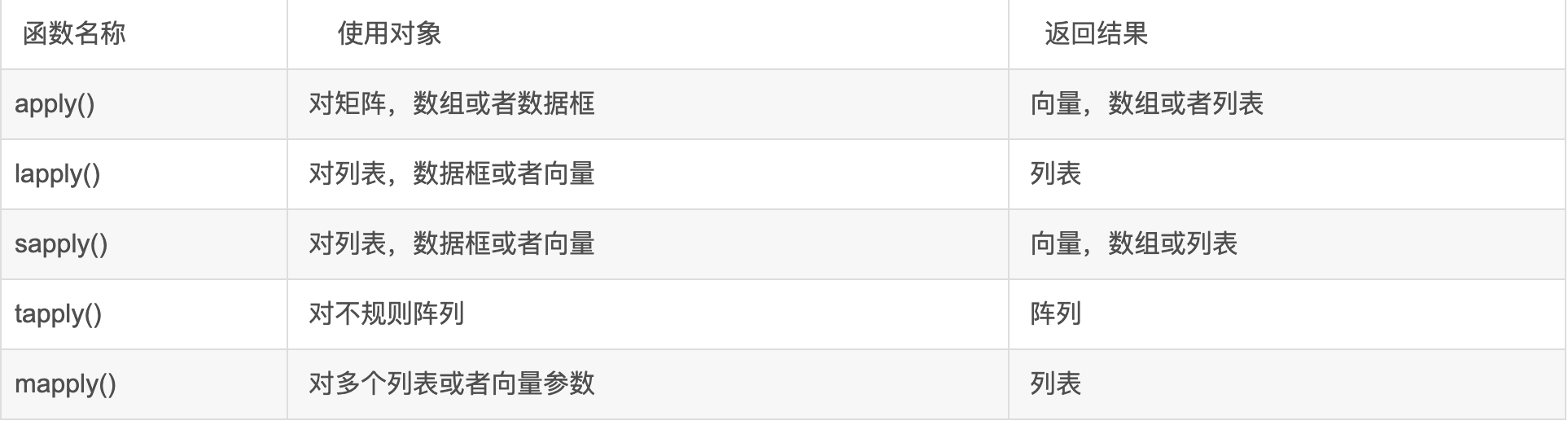
在R语言中,apply系列函数的基本作用是对数组(array,可以是多维)或者列表(list)按照元素或元素构成的子集合进行迭代,并将当前元素或子集合作为参数调用某个指定函数。apply族函数分别有apply函数,tapply函数,lapply函数,mapply函数。每一个函数都有自己的特点,在处理不同类型的数据可以选用相对应的函数。
R 原生英文解释(Apply)
Apply Functions Over Array Margins:
Description
Returns a vector or array or list of values obtained by applying a function to margins of an array or matrix.
Usage
apply(X, MARGIN, FUN, ...)
Arguments
x:
an array, including a matrix.
MARGIN:
a vector giving the subscripts which the function will be applied over. E.g., for a matrix 1 indicates rows, 2 indicates columns, c(1, 2) indicates rows and columns. Where X has named dimnames, it can be a character vector selecting dimension names.
FUN:
the function to be applied: see ‘Details’. In the case of functions like +, %*%, etc., the function name must be backquoted or quoted.
…
optional arguments to FUN.
apply函数(vector)
apply函数只能用于处理矩阵类型的数据,也就是说所有的数据必须是同一类型。因此要使用apply函数的话,需要将数据类型转换成矩阵类型。
apply函数一般有三个参数,第一个参数代表矩阵对象,第二个参数代表要操作矩阵的维度,1表示对行进行处理,2表示对列进行处理。第三个参数就是处理数据的函数。apply会分别一行或一列处理该矩阵的数据。
1 | a<-matrix(1:12,nrow=3) |
使用colMeans(),rowMeans( ),对矩阵的列和行分别求平均值,
rowSums( ),colSums(),,对矩阵的列和行分别求和。
如果数据中NA,那么在求行列的平均值或和的时候,NA所在的行列的计算结果也为NA。
1 | apply(a,1,mean,na.rm=TRUE) |
这样的话,它会自动忽略NA,只计算其他不是NA的值。rowMeans( )中也可以添加na.rm=TRUE参数。效果相同。
1 | #矩阵中添加了一个na |
lapply和sapply函数
lapply和sapply函数可以用于处理列表数据和向量数据(vector/list)
lapply函数得到处理得到的数据类型是列表,
而sapply函数得到处理的数据类型是向量。
这两个函数除了在返回值类型不同外,其他方面基本完全一样。
关于R语言的list数据格式
R中列表(list)类型来保存不同类型的数据。 一个主要目的是提供R分析结果输出包装: 输出一个变量, 这个变量包括回归系数、预测值、残差、检验结果等等一系列不能放到规则形状数据结构中的内容。 实际上,数据框也是列表的一种, 但是数据框要求各列等长, 而列表不要求。
列表可以有多个元素, 但是与向量不同的是, 列表的不同元素的类型可以不同, 比如, 一个元素是数值型向量, 一个元素是字符串, 一个元素是标量, 一个元素是另一个列表。
定义列表用函数list(), 如
1 | rec <- list(name="李明", age=30, |
用typeof()函数判断一个列表, 返回结果为list。 可以用is.list()函数判断某个对象是否列表类型。
列表元素访问
列表的一个元素也可以称为列表的一个“变量”, 单个列表元素必须用两重方括号格式访问,如
1 | rec[[3]] |
列表的单个元素也可以用$格式访问,如
1 | rec$age |
如果使用单重方括号对列表取子集, 结果还是列表而不是列表元素,如
1 | rec[3] |
列表一般都应该有元素名, 元素名可以看成是变量名, 列表中的每个元素看成一个变量。 用names()函数查看和修改元素名。 如
1 | names(rec) |
可以修改列表元素内容。 如
1 | rec[["三科分数"]][2] <- 0 |
直接给列表不存在的元素名定义元素值就添加了新元素, 而且不同于使用向量,对于列表而言这是很正常的做法,比如
1 | rec[["身高"]] <- 178 |
把某个列表元素赋值为NULL就删掉这个元素。 如
1 | rec[["age"]] <- NULL |
在list()函数中允许定义元素为NULL,这样的元素是存在的,如:
1 | li <- list(a=120, b="F", c=NULL); li |
但是,要把已经存在的元素修改为NULL值而不是删除此元素, 或者给列表增加一个取值为NULL的元素, 这时需要用单重的方括号取子集, 这样的子集会保持其列表类型, 给这样的子列表赋值为list(NULL),如:
1 | li["b"] <- list(NULL) |
列表类型转换
用as.list()把一个其它类型的对象转换成列表; 用unlist()函数把列表转换成基本向量。如
1 | li1 <- as.list(1:3) |
1 | li2 <- list(x=1, y=c(2,3)) |
返回列表的函数示例–strsplit()
strsplit()输入一个字符型向量并指定一个分隔符, 返回一个项数与字符型向量元素个数相同的列表, 列表每项对应于字符型向量中一个元素的拆分结果。 如
1 | x <- c("10, 8, 7", "5, 2, 2", "3, 7, 8", "8, 8, 9") |
为了把拆分结果进一步转换成一个数值型矩阵, 可以使用sapply()函数如下:
1 | t(sapply(res, as.numeric)) |
sapply()函数是apply类函数之一, 稍后再详细进行讲解。
https://www.math.pku.edu.cn/teachers/lidf/docs/Rbook/html/_Rbook/prog-type-list.html#p-t-list-as
Test Demo
1 | ```{r} |
tapply函数
- 它通常会有三个参数,第一个参数代表数据,第二个参数表示如何对数据进行分组操作,第三个参数指定每一个分组内应用什么函数。也就是说tapply函数就是把数据按照一定方式分成不同的组,再在每一组数据内进行某种运算。
mapply函数
- mapply函数主要是对多个列表或者向量参数使用函数
————————————————
版权声明:本文为CSDN博主「王亨」的原创文章,遵循CC 4.0 BY-SA版权协议,转载请附上原文出处链接及本声明。
原文链接:https://blog.csdn.net/wzgl__wh/article/details/52207233
demo in DEP
1 | # DEP analysis |
- Post title:R中apply族函数
- Post author:Yuxuan Wu
- Create time:2021-05-06 04:40:35
- Post link:yuxuanwu17.github.io2021/05/06/2021-05-06-R中apply族函数/
- Copyright Notice:All articles in this blog are licensed under BY-NC-SA unless stating additionally.